Publication
-
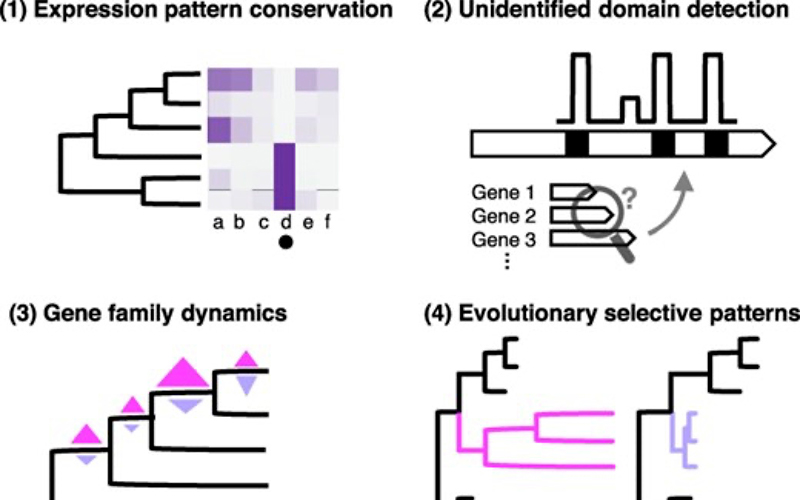 Bessho-Uehara K, Obayashi T.(2025) Evolutionary approaches for narrowing down candidate genes from an unannotated gene list, Plant and Cell Physiology, 66: 287-290.
Bessho-Uehara K, Obayashi T.(2025) Evolutionary approaches for narrowing down candidate genes from an unannotated gene list, Plant and Cell Physiology, 66: 287-290. -
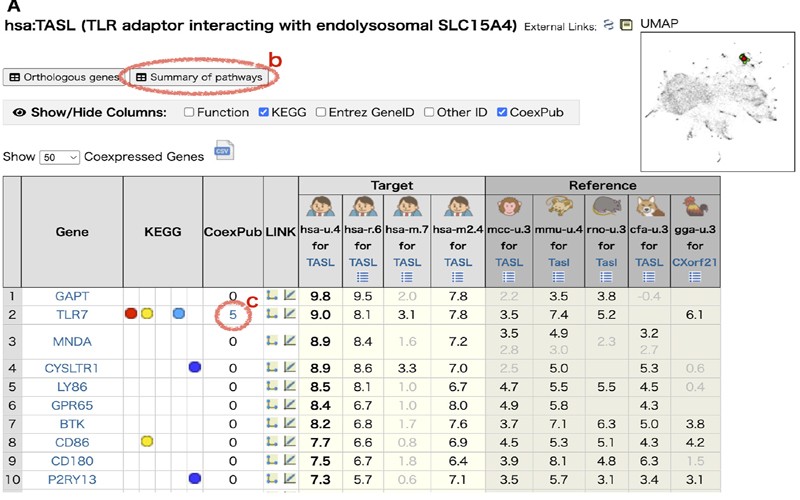 Obayashi T, Kodate S, Hibara H, Kagaya Y, Kinoshita K.(2023) COXPRESdb v8: an animal gene coexpression database navigating from a global view to detailed investigations, Nucleic Acids Research, 51: D80-87.
Obayashi T, Kodate S, Hibara H, Kagaya Y, Kinoshita K.(2023) COXPRESdb v8: an animal gene coexpression database navigating from a global view to detailed investigations, Nucleic Acids Research, 51: D80-87. -
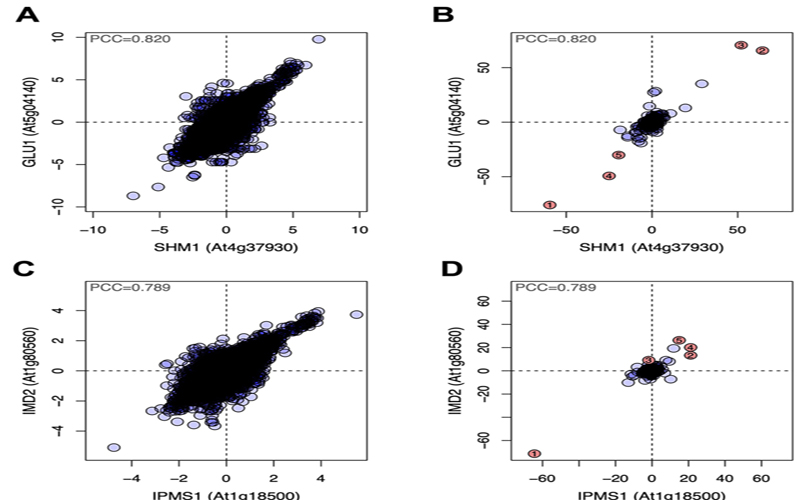 Obayashi T, Hibara H, Kagaya Y, Aoki Y, Kinoshita K. (2022) ATTED-II v11: a plant gene coexpression database using a sample balancing technique by subagging of principal components, Plant Cell Physiology, 63: 869-881.
Obayashi T, Hibara H, Kagaya Y, Aoki Y, Kinoshita K. (2022) ATTED-II v11: a plant gene coexpression database using a sample balancing technique by subagging of principal components, Plant Cell Physiology, 63: 869-881. -
 Obayashi T, Kagaya Y, Aoki Y, Tadaka S, Kinoshita K. (2019) COXPRESdb v7: a gene coexpression database for 11 animal species supported by 23 coexpression platforms for technical evaluation and evolutionary inference, Nucleic Acids Research, 47: D55-D62.
Obayashi T, Kagaya Y, Aoki Y, Tadaka S, Kinoshita K. (2019) COXPRESdb v7: a gene coexpression database for 11 animal species supported by 23 coexpression platforms for technical evaluation and evolutionary inference, Nucleic Acids Research, 47: D55-D62. -
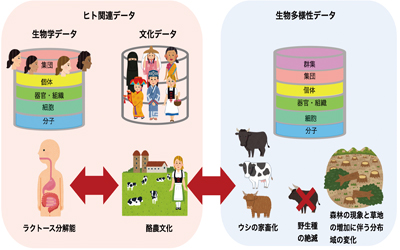 松前ひろみ, 神保宇嗣, 仲里猛留, 畠山剛臣, 大林武. (2022) 生物多様性と文化へと繋がるバイオインフォマティクス, JSBi Bioinformatics Review, 3: 88-114.
松前ひろみ, 神保宇嗣, 仲里猛留, 畠山剛臣, 大林武. (2022) 生物多様性と文化へと繋がるバイオインフォマティクス, JSBi Bioinformatics Review, 3: 88-114.
Presentation
2025
-
 Dilokkalayakul J, Kitamura A, Obayashi T. Context-Aware Marine Plankton Classification with Multimodal Large Language Model and Retrieval-Augmented Generation Reasoning, 11th EAFES International Congress, 2025年7月21日, University of Tokyo.
Dilokkalayakul J, Kitamura A, Obayashi T. Context-Aware Marine Plankton Classification with Multimodal Large Language Model and Retrieval-Augmented Generation Reasoning, 11th EAFES International Congress, 2025年7月21日, University of Tokyo. -
 Obayashi T. Monitoring Coastal Plankton Communities in Northeast Japan through Metagenomics and Epigenomics in the PlanDyO Project, 11th EAFES International Congress, 2025年7月8日, University of Tokyo.
Obayashi T. Monitoring Coastal Plankton Communities in Northeast Japan through Metagenomics and Epigenomics in the PlanDyO Project, 11th EAFES International Congress, 2025年7月8日, University of Tokyo. -
 Dilokkalayakul J. the IMBC Award Best Oral Presentation, International Marine Biotechnology Conference (IMBC), 2025年7月8日, Brest, FRANCE.
Dilokkalayakul J. the IMBC Award Best Oral Presentation, International Marine Biotechnology Conference (IMBC), 2025年7月8日, Brest, FRANCE. -
 Dilokkalayakul J, Kitamura A, Obayashi T. Automated Taxonomic Identification of Marine Plankton Using a Large-Language-Model and Retrieval-Augmented-Generation Framework, International Marine Biotechnology Conference (IMBC), 2025年7月8日, Brest, FRANCE.
Dilokkalayakul J, Kitamura A, Obayashi T. Automated Taxonomic Identification of Marine Plankton Using a Large-Language-Model and Retrieval-Augmented-Generation Framework, International Marine Biotechnology Conference (IMBC), 2025年7月8日, Brest, FRANCE. -
 Obayashi T. PlanDyO: A Long-Term Meta-Epigenomic Platform to Elucidate Marine Plankton Dynamics in Onagawa Bay, Sanriku Coast, International Marine Biotechnology Conference (IMBC), 2025年7月8日, Brest, FRANCE.
Obayashi T. PlanDyO: A Long-Term Meta-Epigenomic Platform to Elucidate Marine Plankton Dynamics in Onagawa Bay, Sanriku Coast, International Marine Biotechnology Conference (IMBC), 2025年7月8日, Brest, FRANCE. -
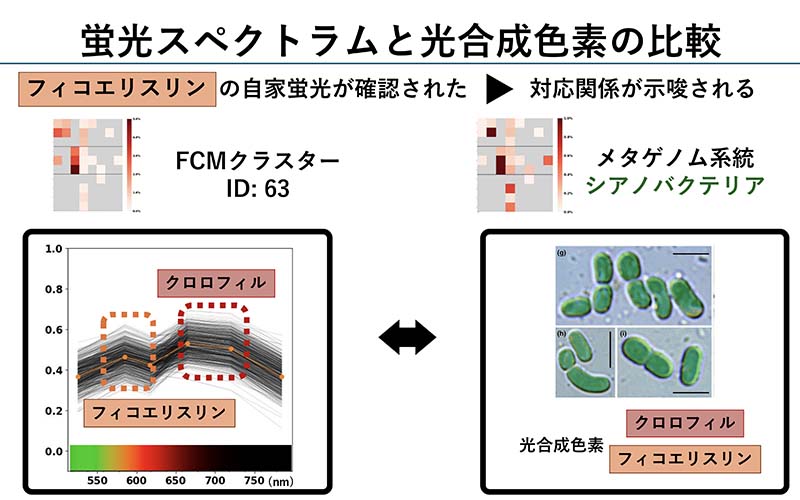 邊見匠馬, 北村茜, 藤井豊展, 池田実, 大林武. 海洋性プランクトンのマルチモーダル解析における相関ネットワークの評価, 情報処理学会第153回MPS・第82回BIO合同研究発表会, 2025年6月23日, 琉球大学.
邊見匠馬, 北村茜, 藤井豊展, 池田実, 大林武. 海洋性プランクトンのマルチモーダル解析における相関ネットワークの評価, 情報処理学会第153回MPS・第82回BIO合同研究発表会, 2025年6月23日, 琉球大学. -
 Obayashi T. PlanDyO: Monitoring Marine Plankton Dynamics Using Meta- and Epi-genomics in Northeast Japan, AIMEC Workshop "Ocean environmental change and its impact on ecosystems" 2025年6月23日, Scripps Institution of Oceanography.
Obayashi T. PlanDyO: Monitoring Marine Plankton Dynamics Using Meta- and Epi-genomics in Northeast Japan, AIMEC Workshop "Ocean environmental change and its impact on ecosystems" 2025年6月23日, Scripps Institution of Oceanography. -
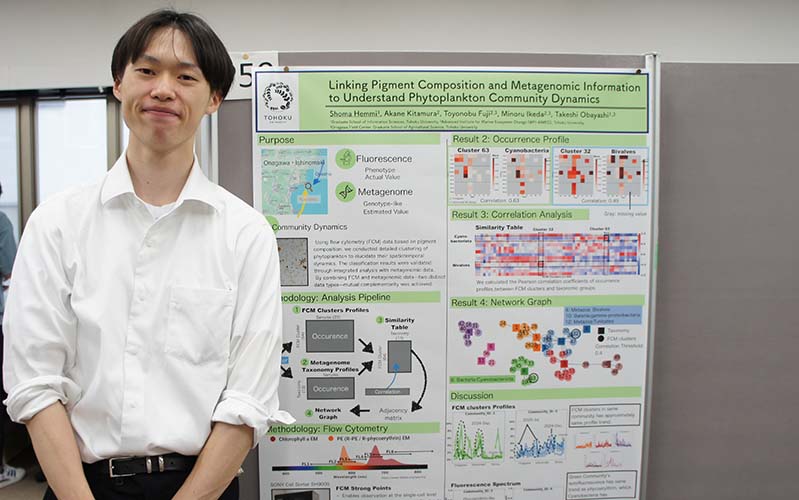 Hemmi S, Kitamura A, Fujii T, Ikeda M, Obayashi T. Linking Pigment Composition and Metagenomic Information to Understand Phytoplankton Community Dynamics, Earth, Sea and Sky XI International Workshop, 2025年6月4日, 東北大学.
Hemmi S, Kitamura A, Fujii T, Ikeda M, Obayashi T. Linking Pigment Composition and Metagenomic Information to Understand Phytoplankton Community Dynamics, Earth, Sea and Sky XI International Workshop, 2025年6月4日, 東北大学. -
 Obayashi T. Meta-epigenomic monitoring of marine plankton dynamics in current-influenced coastal ecosystems of northeastern Japan, Earth, Sea and Sky XI International Workshop, 2025年6月4日, 東北大学.
Obayashi T. Meta-epigenomic monitoring of marine plankton dynamics in current-influenced coastal ecosystems of northeastern Japan, Earth, Sea and Sky XI International Workshop, 2025年6月4日, 東北大学. -
 Dilokkalayakul J, Kitamura A, Obayashi T. Semantic Plankton: Multimodal LLMs and RAG System for Automated Marine Plankton Analysis, Earth, Sea and Sky XI International Workshop, 2025年6月3日, 東北大学.
Dilokkalayakul J, Kitamura A, Obayashi T. Semantic Plankton: Multimodal LLMs and RAG System for Automated Marine Plankton Analysis, Earth, Sea and Sky XI International Workshop, 2025年6月3日, 東北大学. -
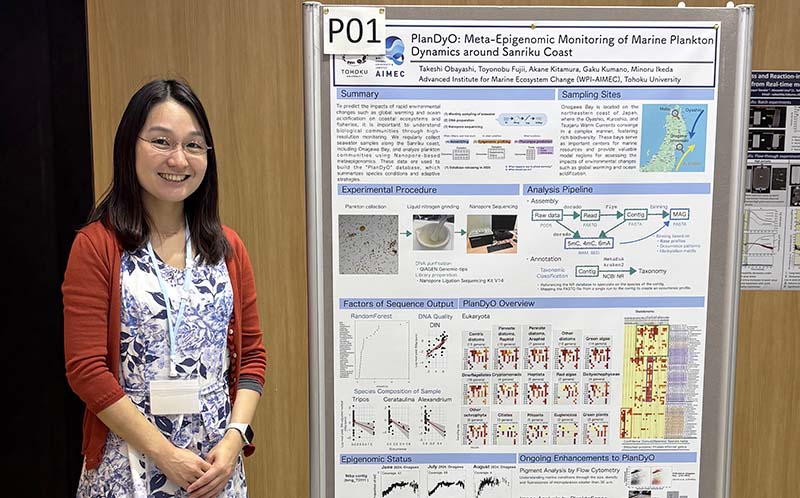 Obayashi T, Fujii T, Kitamura A, Kumano G, Ikeda M. Meta-Epigenomic Monitoring of Marine Plankton Dynamics around Sanriku Coast, Earth, Sea and Sky XI International Workshop, 2025年6月3日, 東北大学.
Obayashi T, Fujii T, Kitamura A, Kumano G, Ikeda M. Meta-Epigenomic Monitoring of Marine Plankton Dynamics around Sanriku Coast, Earth, Sea and Sky XI International Workshop, 2025年6月3日, 東北大学. -
 大林武. 地球環境変化に応答する沿岸生態系の理解に向けた長期メタゲノム解析, TCPAIシンポジウム2025, 2025年6月3日, 東北大学災害科学国際研究所.
大林武. 地球環境変化に応答する沿岸生態系の理解に向けた長期メタゲノム解析, TCPAIシンポジウム2025, 2025年6月3日, 東北大学災害科学国際研究所. -
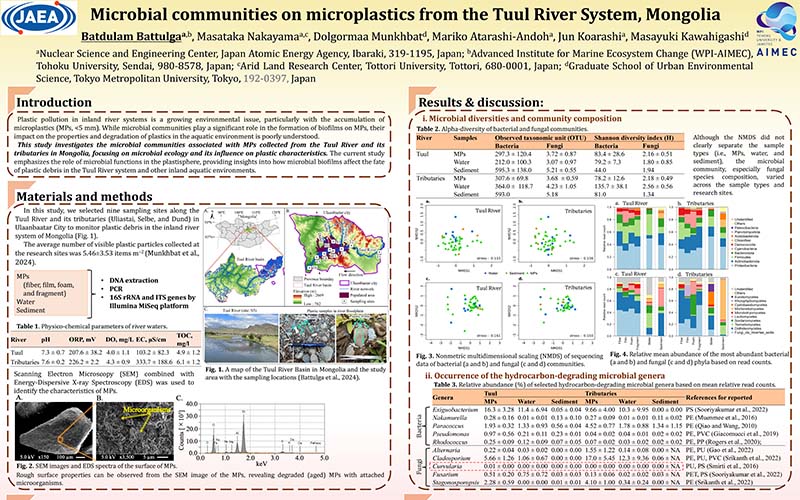 Batudulum B. Microbial Communities on Microplastics from the Tuul River System, Mongolia, Japan Geoscience Union Meeting 2025, 2025年5月29日, 幕張メッセ.
Batudulum B. Microbial Communities on Microplastics from the Tuul River System, Mongolia, Japan Geoscience Union Meeting 2025, 2025年5月29日, 幕張メッセ. -
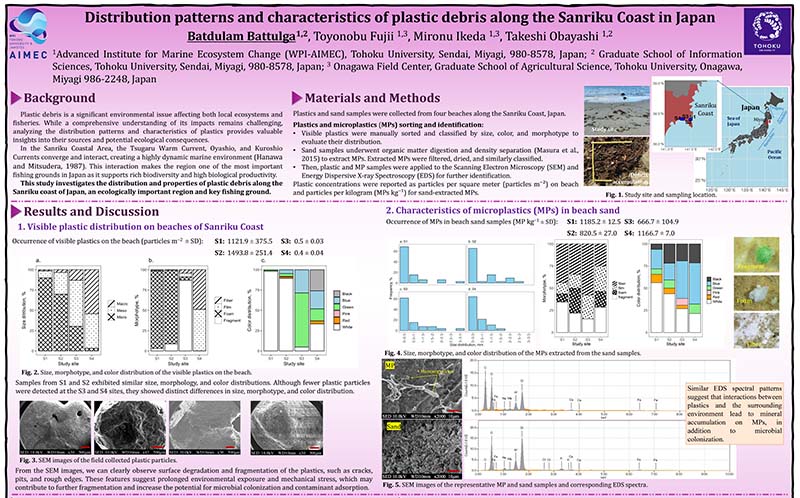 Batudulum B. Distribution Patterns and Characteristics of Plastic Debris Along the Sanriku Coast in Japan, Japan Geoscience Union Meeting 2025, 2025年5月26日, 幕張メッセ.
Batudulum B. Distribution Patterns and Characteristics of Plastic Debris Along the Sanriku Coast in Japan, Japan Geoscience Union Meeting 2025, 2025年5月26日, 幕張メッセ. -
 Batudulum B. Plastic-Associated Radiocesium and Microbial Communities in Coastal Rivers of Fukushima, Japan, Japan Geoscience Union Meeting 2025, 2025年5月26日, 幕張メッセ.
Batudulum B. Plastic-Associated Radiocesium and Microbial Communities in Coastal Rivers of Fukushima, Japan, Japan Geoscience Union Meeting 2025, 2025年5月26日, 幕張メッセ. -
 大林武, 藤井豊展, 北村茜, 熊野岳, 池田実 沿岸プランクトンの多様性と適応戦略を俯瞰するメタエピゲノム解析データベース PlanDyO, 生物多様性のDNA情報学 2025, 2025年4月24日, 国立遺伝学研究所.
大林武, 藤井豊展, 北村茜, 熊野岳, 池田実 沿岸プランクトンの多様性と適応戦略を俯瞰するメタエピゲノム解析データベース PlanDyO, 生物多様性のDNA情報学 2025, 2025年4月24日, 国立遺伝学研究所. -
 Kitamura A. PlanDyO: Meta-Epigenomic Monitoring of Marine Plankton Dynamics in Onagawa and Mutsu Bay, Graduate School of Agricultural Science, Tohoku University The 22nd International Symposium on Integrated Field Science, 2025年3月14日, Tohoku University.
Kitamura A. PlanDyO: Meta-Epigenomic Monitoring of Marine Plankton Dynamics in Onagawa and Mutsu Bay, Graduate School of Agricultural Science, Tohoku University The 22nd International Symposium on Integrated Field Science, 2025年3月14日, Tohoku University. -
 Obayashi T. ATTED-II Version 12.0: A Plant Gene Coexpression Database for Comparative Analysis Across Flowering Plants, 第66回日本植物生理学会年会, 2025年3月14日, 金沢大学.
Obayashi T. ATTED-II Version 12.0: A Plant Gene Coexpression Database for Comparative Analysis Across Flowering Plants, 第66回日本植物生理学会年会, 2025年3月14日, 金沢大学. -
 Batudulum B. Dynamics and functions of microbial communities and biofilm characteristics in the Plastisphere in temperate coastal environments, Workshop for Collaborative Research of AIMEC, 2025年3月4日, University of Hawaii at Manoa, USA.
Batudulum B. Dynamics and functions of microbial communities and biofilm characteristics in the Plastisphere in temperate coastal environments, Workshop for Collaborative Research of AIMEC, 2025年3月4日, University of Hawaii at Manoa, USA. -
 Dilokkalayakul J. Advancing Plankton Monitoring: Automatic Image Analysis with PlanktoScope in the PlanDyO Project, Workshop for Collaborative Research of AIMEC, 2025年3月4日, University of Hawaii at Manoa, USA.
Dilokkalayakul J. Advancing Plankton Monitoring: Automatic Image Analysis with PlanktoScope in the PlanDyO Project, Workshop for Collaborative Research of AIMEC, 2025年3月4日, University of Hawaii at Manoa, USA. -
 Obayashi T. PlanDyO: Long-term Meta-Epigenomic Monitoring Platform to Elucidate Marine Plankton Dynamics along the Sanriku Coast, Workshop for Collaborative Research of AIMEC, 2025年3月4日, University of Hawaii at Manoa, USA.
Obayashi T. PlanDyO: Long-term Meta-Epigenomic Monitoring Platform to Elucidate Marine Plankton Dynamics along the Sanriku Coast, Workshop for Collaborative Research of AIMEC, 2025年3月4日, University of Hawaii at Manoa, USA. -
Batudulum B. Microplastics and biofilm dynamics in the Kuroshio Extension Region of the Northwest Pacific Ocean, the 2025 “Kaimei” cruise organized by JAMSTEC, 2025年1月31日, JAMSTEC横須賀本部.
-
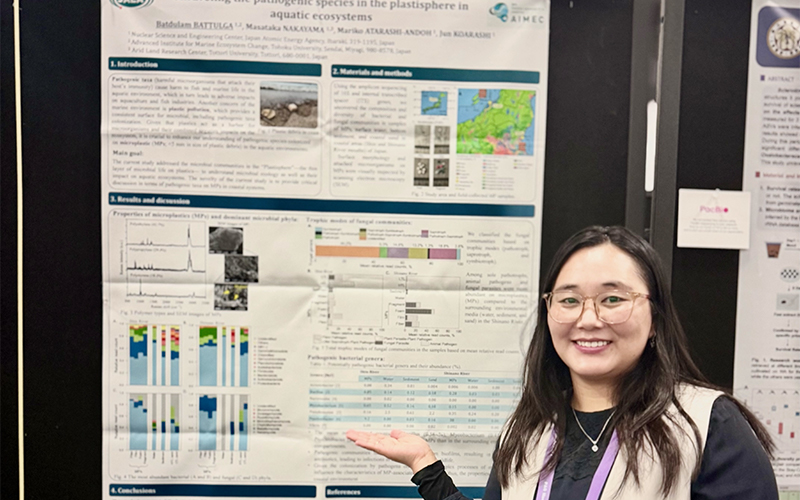 Batudulum B. Unraveling the pathogenic species in the plastisphere in aquatic ecosystems, Plant & Animal Genome Conference 2025, 2025年1月13日, The Town & Country Resort and Conference Center, San Diego, USA.
Batudulum B. Unraveling the pathogenic species in the plastisphere in aquatic ecosystems, Plant & Animal Genome Conference 2025, 2025年1月13日, The Town & Country Resort and Conference Center, San Diego, USA. -
 Obayashi T. Long-Term Meta-Epigenomic Monitoring Platform to Elucidate Marine Plankton Dynamics in Onagawa Bay, Sanriku Coast, Plant & Animal Genome Conference 2025, 2025年1月13日, The Town & Country Resort and Conference Center, San Diego, USA.
Obayashi T. Long-Term Meta-Epigenomic Monitoring Platform to Elucidate Marine Plankton Dynamics in Onagawa Bay, Sanriku Coast, Plant & Animal Genome Conference 2025, 2025年1月13日, The Town & Country Resort and Conference Center, San Diego, USA.
2024
-
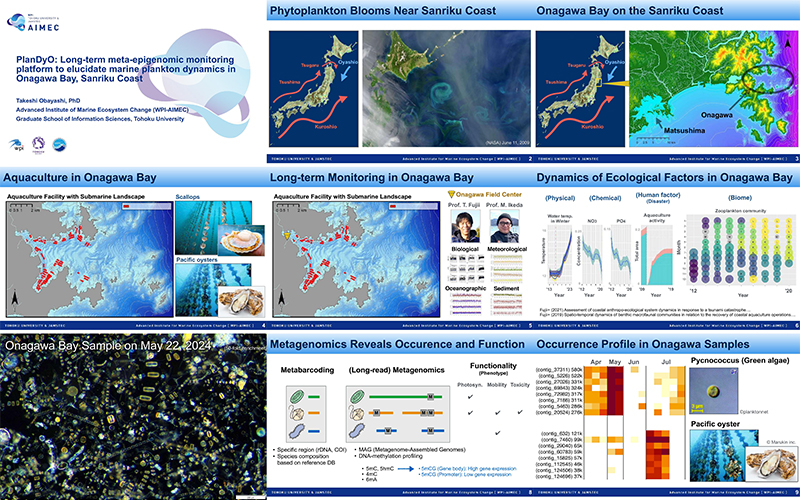 Obayashi T. PlanDyO: Long-term Meta-Epigenomic Monitoring Platform to Elucidate Marine Plankton Dynamics in Onagawa Bay, Sanriku Coast, the 2nd International Scientific Symposium of CSK-2, 2024年11月24日, Tohoku University.
Obayashi T. PlanDyO: Long-term Meta-Epigenomic Monitoring Platform to Elucidate Marine Plankton Dynamics in Onagawa Bay, Sanriku Coast, the 2nd International Scientific Symposium of CSK-2, 2024年11月24日, Tohoku University. -
 Obayashi T. ATTED-II Version 12.0: Plant Gene Coexpression Database with Enhanced Species Comparison for Supporting Flowering Plants, 1st Asia & Pacific Bioinformatics Joint Conference, 2024年10月24日, 那覇文化芸術劇場なはーと.
Obayashi T. ATTED-II Version 12.0: Plant Gene Coexpression Database with Enhanced Species Comparison for Supporting Flowering Plants, 1st Asia & Pacific Bioinformatics Joint Conference, 2024年10月24日, 那覇文化芸術劇場なはーと. -
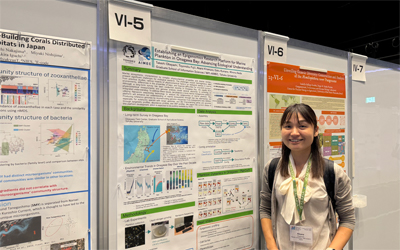 Obayashi T, Fujii T, Kitamura A, Kumano G, Ikeda M. Establishing an Epigenomics Research Platform for Marine Plankton in Onagawa Bay:Advancing Ecological Understanding, 1st Asia & Pacific Bioinformatics Joint Conference, 2024年10月24日, 那覇文化芸術劇場なはーと.
Obayashi T, Fujii T, Kitamura A, Kumano G, Ikeda M. Establishing an Epigenomics Research Platform for Marine Plankton in Onagawa Bay:Advancing Ecological Understanding, 1st Asia & Pacific Bioinformatics Joint Conference, 2024年10月24日, 那覇文化芸術劇場なはーと. -
 大林武. 植物の遺伝子共発現データベースATTED-II version 12.0, トーゴーの日シンポジウム2024, 2024年10月5日, 品川ザ・グランドホール.
大林武. 植物の遺伝子共発現データベースATTED-II version 12.0, トーゴーの日シンポジウム2024, 2024年10月5日, 品川ザ・グランドホール. -
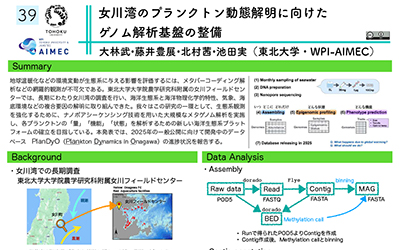 大林武, 藤井豊展, 北村茜, 池田実. 女川湾のプランクトン動態解明に向けたゲノム解析基盤の整備, トーゴーの日シンポジウム2024, 2024年10月5日, 品川ザ・グランドホール.
大林武, 藤井豊展, 北村茜, 池田実. 女川湾のプランクトン動態解明に向けたゲノム解析基盤の整備, トーゴーの日シンポジウム2024, 2024年10月5日, 品川ザ・グランドホール. -
 大林武. 植物の共発現データベースATTED-IIにおける種間比較機能強化の取り組み, DICP研究交流会, 2024年10月4日, JST東京本部.
大林武. 植物の共発現データベースATTED-IIにおける種間比較機能強化の取り組み, DICP研究交流会, 2024年10月4日, JST東京本部. -
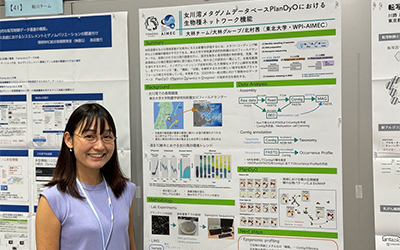 北村茜. 女川湾メタゲノムデータベースPlanDyOにおける生物種ネットワーク機能, DICP研究交流会, 2024年10月4日, JST東京本部.
北村茜. 女川湾メタゲノムデータベースPlanDyOにおける生物種ネットワーク機能, DICP研究交流会, 2024年10月4日, JST東京本部. -
 邊見匠馬, 笠原大輔, 森山祥太, 中嶋正道. 画像分類AIによるイワナ地域個体群分類の試み, 令和6年度日本水産学会秋季大会, 2024年9月25日, 京都大学.
邊見匠馬, 笠原大輔, 森山祥太, 中嶋正道. 画像分類AIによるイワナ地域個体群分類の試み, 令和6年度日本水産学会秋季大会, 2024年9月25日, 京都大学. -
 Nishikawa Y, Obayashi T, Matsumae H. Visualizing human impact on plant transmission from linguistic data, Cultural Evolution Society Conference 2024, 9-11 Sep 2024, Durham University (UK).
Nishikawa Y, Obayashi T, Matsumae H. Visualizing human impact on plant transmission from linguistic data, Cultural Evolution Society Conference 2024, 9-11 Sep 2024, Durham University (UK). -
 内田克哉, 大林武. 生命科学分野における電界撹拌技術を用いたin situ hybridization法の迅速化の検討, 第14回迅速免疫染色研究会, 2024年9月7日, 旭川医科大学病院.
内田克哉, 大林武. 生命科学分野における電界撹拌技術を用いたin situ hybridization法の迅速化の検討, 第14回迅速免疫染色研究会, 2024年9月7日, 旭川医科大学病院. -
 Lasserre E. Unraveling Gene Expression Evolution in Subgenomes of Allopolyploid Wheat, TOHOKU University 2023-2024 Spring Semester COLABS, 7 Aug 2024, Tohoku University.
Lasserre E. Unraveling Gene Expression Evolution in Subgenomes of Allopolyploid Wheat, TOHOKU University 2023-2024 Spring Semester COLABS, 7 Aug 2024, Tohoku University. -
 Obayashi T. Meta-Epigenomic Insights into Marine Plankton Dynamics in Onagawa Bay, 10th Annual Data Science in Engineering and Life Sciences Symposium, 6 Aug 2024, Case Western Reserve University (Cleveland, OH ).
Obayashi T. Meta-Epigenomic Insights into Marine Plankton Dynamics in Onagawa Bay, 10th Annual Data Science in Engineering and Life Sciences Symposium, 6 Aug 2024, Case Western Reserve University (Cleveland, OH ). -
 大林武. 変わりゆく海:海洋ビッグデータ解析の挑戦, Public lecture in Open Campus 2024, 2024年7月31日, 東北大学.
大林武. 変わりゆく海:海洋ビッグデータ解析の挑戦, Public lecture in Open Campus 2024, 2024年7月31日, 東北大学. -
 内田克哉. 電解撹拌装置を用いた組織染色の迅速化, 日本動物学会・令和6年度東北支部大会, 2024年7月27日, 山形大学.
内田克哉. 電解撹拌装置を用いた組織染色の迅速化, 日本動物学会・令和6年度東北支部大会, 2024年7月27日, 山形大学. -
 Lasserre E. Unraveling Gene Expression Evolution in Subgenomes of Allopolyploid Wheat, 4th GPDS Science Cafe , 22 July 2024, Tohoku University.
Lasserre E. Unraveling Gene Expression Evolution in Subgenomes of Allopolyploid Wheat, 4th GPDS Science Cafe , 22 July 2024, Tohoku University. -
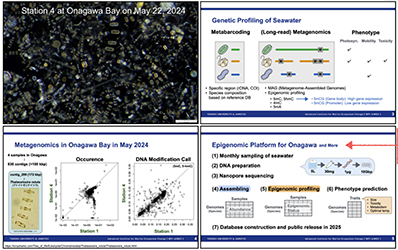 Obayashi T. Establishing an Epigenomics Research Platform for Marine Plankton: From Individual Species Analyses to Ecological Modeling., 1st AIMEC colloquium, 18 July 2024, Tohoku University.
Obayashi T. Establishing an Epigenomics Research Platform for Marine Plankton: From Individual Species Analyses to Ecological Modeling., 1st AIMEC colloquium, 18 July 2024, Tohoku University. -
 大林武. 非モデル植物のための遺伝子ネットワーク情報活用基盤, 2024年度 統合化推進プログラム サイトビジット, 2024年7月4日, 東北大学.
大林武. 非モデル植物のための遺伝子ネットワーク情報活用基盤, 2024年度 統合化推進プログラム サイトビジット, 2024年7月4日, 東北大学. -
 Obayashi T. Establishing an Epigenomics Research Platform for Marine Plankton Through Long-Term Monitoring in Onagawa Bay, AIMEC Asamushi Workshop, 17 June 2024, 道の駅ゆ〜さ浅⾍.
Obayashi T. Establishing an Epigenomics Research Platform for Marine Plankton Through Long-Term Monitoring in Onagawa Bay, AIMEC Asamushi Workshop, 17 June 2024, 道の駅ゆ〜さ浅⾍. Obayashi T, Ikeda M, Fujii T, Hamabata T. Onagawa Bay: A Model for Advanced Coastal Ecosystem Research Through Genomics and Data Integration, Japan Geoscience Union Meeting 2024, 27 May 2024, MAKUHARI MESSE.
Obayashi T, Ikeda M, Fujii T, Hamabata T. Onagawa Bay: A Model for Advanced Coastal Ecosystem Research Through Genomics and Data Integration, Japan Geoscience Union Meeting 2024, 27 May 2024, MAKUHARI MESSE. Obayashi T. Enhancing ATTED-II Database for Diverse Plant Species Research, 第65回日本植物生理学会年会, 2024年3月17日, 神戸国際会議場.西川有理, 松前ひろみ, 大林武. Visualizing human impact on plant transmission from linguistic data, 第71回日本生態学会大会, 2024年3月13日, 横浜国立大学.
Obayashi T. Enhancing ATTED-II Database for Diverse Plant Species Research, 第65回日本植物生理学会年会, 2024年3月17日, 神戸国際会議場.西川有理, 松前ひろみ, 大林武. Visualizing human impact on plant transmission from linguistic data, 第71回日本生態学会大会, 2024年3月13日, 横浜国立大学. Obayashi T. Coastal Ecosystem Services Research Unit, WPI-AIMEC Kick-off Symposium, 8 Mar 2024, Tohoku University.
Obayashi T. Coastal Ecosystem Services Research Unit, WPI-AIMEC Kick-off Symposium, 8 Mar 2024, Tohoku University. 西川有理, 松前ひろみ, 大林武. 植物の伝播における人間の影響を言語データから可視化する, 東海大学 マイクロ・ナノ啓発会, 2024年2月29日, 東海大学.
西川有理, 松前ひろみ, 大林武. 植物の伝播における人間の影響を言語データから可視化する, 東海大学 マイクロ・ナノ啓発会, 2024年2月29日, 東海大学.2023
-
Obayashi T, Ikeda M. Kinship Analysis on wild populations of olive flounder, RIEC International Symposium / Symposium of Yotta Informatics Research Platform for Yotta-Scale Data Science 2023, 9 Mar 2023, online.
-
Obayashi T. Subagging of Principal Components for Sample Balancing: Building a Condition-Independent Gene Coexpression Resource from Public Transcriptome Data, Function COSI, ISMB/ECCB 2023 (Intelligent Systems For Molecular Biology / European Conference On Conputational Biology) 26 Jul 2023, Lyon, France.
-
大林武. 非モデル植物のための遺伝子ネットワーク情報活用基盤, トーゴーの日シンポジウム, 2023年10月5日, 日本科学未来館.
-
大林武. 共発現データベースATTED-IIにおける種間比較の取り組み, 統合化推進プログラム(DICP)研究交流会, 2023年10月5日, 日本科学未来館.
-
火原日美子. 共発現データベースATTED-IIにおけるバージョン管理, 統合化推進プログラム(DICP)研究交流会, 2023年10月5日, 日本科学未来館.
-
大林武, 火原日美子, 木下賢吾. Genome-Scale Gene Coexpression Visualization Tools in Gene Coexpression Database COXPRESdb, IIBMP2023, 2023年9月7日, 柏の葉カンファレンスセンター.
-
西川有理, 松前ひろみ, 大林武. The transmission of plant names across languages can be visualized from open data, IIBMP2023, 2023年9月7日, 柏の葉カンファレンスセンター.
-
大林武. 遺伝子共発現データベースATTED-IIにおける種固有の共発現情報の導出と利用の展望, 第1回北海道バイオ"Mix up", 2023年8月8日, 北海道大学.
-
大林武. Platform of Gene Network Information for Non-model Plants, 統合化推進プログラム(DICP)キックオフ・ミーティング, 2023年5月24, online.
-
Tamer N. An interdisciplinary investigation of categorical call distinction in meerkat vocalisations, 2nd GP-DS Data Science Cafe, 25 Mar 2023, Tohoku University.
-
Obayashi T. Gene coexpression resource for predicting all metabolic pathways in a species, 2nd GP-DS Data Science Cafe, 25 Mar 2023, Tohoku University.
-
大林武. The coexpression map provides an entry point for exploring gene coexpression space in ATTED-Il v11, 第64回日本植物生理学会年会, 2023年3月15日, 東北大学.
-
Obayashi T. ATTED-II; a plant gene coexpression database with challenges for inter-species comparison, MPIPZ Special Seminar, 23 Jan 2023, Max Planck Institute for Plant Breeding Research.
2022
-
Obayashi T. Gene coexpression network for inter-species comparison based on publicly available transcriptome data, BEEES (Behaviour, Ecology, Environment and Evolution Seminar), 24 Feb 2022, University of Zurich, Zurich, Switzerland.
-
Obayashi T, Ikeda M. Kinship Analysis on wild populations of olive flounder, Symposium of Yotta Informatics - Research Platform for Yotta-Scale Data Science 2022, 17 Mar 2022, Tohoku University. (online)
-
Obayashi T, Aoki Y. Gene-to-gene Spearman correlation using the sample principal component scores is a simple sample-balancing methodology for gene coexpression calculation, 日本植物生理学会年会, 2022年3月23日, つくば大学. (online)
-
大林武, 安藤大樹, 池田実. 野生ヒラメの血縁推定における尤度比のパーミュテーション検定, 第70回情報処理学会バイオ情報学研究会 (SIGBIO70), 2022年6月29日, 琉球大学.
-
大林武, 安藤大樹, 池田実. マイクロサテライトを用いた野生ヒラメの血縁推定における尤度比のパーミュテーション検定, 第24回日本進化学会年大会, 2022年8月5日, 沼津.
-
Obayashi T, Kodate S, Hibara H, Kagaya Y, Kinoshita K. 遺伝子共発現データベースCOXPRESdb, トーゴーの日2022, 2022年10月5日, online.
-
田中瑞樹, 大林武, 吉田聡子. 寄生植物コシオガマの遺伝子共発現ネットワーク解析, 日本植物学会第86回大会, 2022年9月17日, 京都府立大学.
-
田中瑞樹, 大林武, 吉田聡子. 寄生植物コシオガマの遺伝子共発現ネットワーク解析, 第11回近畿植物学会講演会, 2022年11月19日, 大阪大学.
-
大久保叡一, 青木裕一, 大林武, 永野惇, 岩山幸治. Variational Autoencoderを用いた組織種による遺伝子発現データの分離, 2022年度統計関連学会連合大会, 2022年9月7日, 成蹊大学.
